启动子的分析和预测
一、摘要
- 加深对基因启动子的理解和认知;
- 学会如何获取已知基因的启动子序列数据;
- 熟悉不同启动子分析软件的使用及其适用范围;
- 学会设计启动子克隆引物。
- 熟悉EPD和TransFac数据库的使用;
- 学会使用已知的启动子和转录因子TransFac的HMM模型,并能够独立编程,利用该HMM模型来计算鉴别未知启动子
二、材料和方法
1、硬件平台
处理器:Intel(R) Core(TM)i7-4710MQ CPU @ 2.50GHz
安装内存(RAM):16.0GB
2、系统平台
Windows 8.1、Ubuntu
3、软件平台
【1】Primer-BLAST
【2】Softberry系列工具
【3】Promoter 2.0
【4】BDGP
【5】Cister
【6】NEBcutter
4、数据库资源
NCBI数据库:https://www.ncbi.nlm.nih.gov/
UCSC数据库:http://genome.ucsc.edu/
5、研究对象
人类谷胱甘肽硫转移酶M1的promoter区域
三、结果
基因启动子序列的获取
选择基因:谷胱甘肽硫转移酶M1(GSTM1)
概况:当携带风险基因型时,对环境毒素和致癌物质的敏感性提高,易发生DNA突变和染色体畸变,患白血病的风险因而显著增加。
首先进入UCSC genome browser 查看GSTM1上游5kb范围内有无其他基因。发现该基因的上游存在同一家族的GSTM2,所以promoter大概只有3kb。

图表 1UCSC genome browser
接下来进入Genbank,搜索GSTM1,查看该基因在基因组中的定位和基因结构。

图表 2查看基因定位和结构
打开该基因的序列信息,获取该基因的启动子序列(包含exon1)


Neural Network Promoter Prediction
进入BDGP: Neural Network Promoter Prediction网站http://www.fruitfly.org/seq_tools/promoter.html,进行启动子预测

图表 3 BDGP: Neural Network Promoter Prediction网站
一共预测出来3个启动子(这个网站预测出来的promoter都是50bp)

Promoter 2.0 Prediction
使用Promoter 2.0 Prediction Server http://www.cbs.dtu.dk/services/Promoter/
进行启动子预测,也是一共预测出来3个启动子

图表 5Promoter 2.0预测结果
Softberry预测
TSSW、TSSP、TSSG、FPROM都是softberry提供的启动子预测工具,进入
官网(http://www.softberry.com/),然后点击service即可,启动子预测工具网址:
http://www.softberry.com/berry.phtml?topic=index&group=programs&subgroup=promoter
TSSW
TSSW具体网址如下(http://www.softberry.com/berry.phtml?topic=tssw&group=programs&subgroup=promoter),输入序列进行预测即可。TSSW并没有预测出来promoter区域。

图表 6TSSW预测结果
TSSP
TSSP具体网址如下(http://www.softberry.com/berry.phtml?topic=tssp&group=programs&subgroup=promoter),输入序列进行预测即可。共计预测出来一个promoter区域。

图表 7 TSSP预测结果
TSSG
TSSG具体网址如下(http://www.softberry.com/berry.phtml?topic=tssg&group=programs&subgroup=promoter),输入序列进行预测即可。TSSG并没有预测出来promoter区域。

图表 8TSSG预测结果
FPROM
FPROM具体网址如下(http://www.softberry.com/berry.phtml?topic=fprom&group=programs&subgroup=promoter),输入序列进行预测即可。FPROM并没有预测出来promoter区域。

图表 9FPROM预测结果
Cister
Transcription Elements预测平台:Cis-element Cluster Finder
https://zlab.bu.edu/~mfrith/cister.shtml
由于序列只有3kb,默认参数预测出来的转录元件太少,将average distance between clusters参数由默认的3w修改为3k,最有可能的结果还是NF-1

图表 10Cister预测结果
Match
转录因子预测集合网站http://gene-regulation.com/pub/programs.html (需要注册)
具体网址http://gene-regulation.com/cgi-bin/pub/programs/match/bin/match.cgi

图表 11Match预测结果
AliBaba 2.1
转录因子预测集合网站http://gene-regulation.com/pub/programs.html (需要注册)
具体网址http://gene-regulation.com/pub/programs/alibaba2/index.html
预测出来一大堆,下面仅截取其中一部分。

图表 12AliBaba预测结果
基因结构绘图
虽然使用了6个promoter预测在线平台,但是只有3个平台预测出promoter。
利用在线平台processon绘制基因结构图

图表 13基因结构图
接下来大致将预测结果分为4个区域,将引物设计的范围同时绘制出来(箭头为引物)
PrimerBlast引物设计

图表 14引物结构
先使用默认参数进行尝试,设置好Forward primer和Reverse primer的Range,再将PCR product size最大值调成整段序列的长度3005,同时# of primers to return参数调整为1,以方便截图。
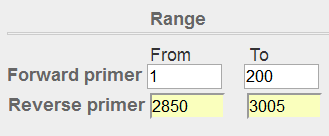
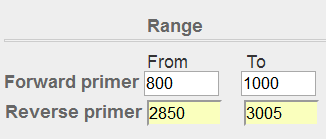
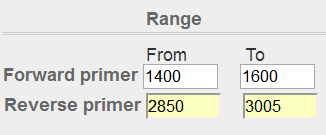
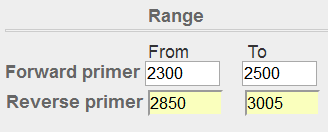
图表 15引物位置

图表 16初步设置参数
然而由于有重复序列,经过repeat filtering,不会跑出来引物,只能将参数调宽松。

图表 17默认参数结果
在Advanced parameters的Primer Parameters栏目,将Repeat filter关掉,同样可以看出来GC含量和TM值也筛选掉很多引物,在Internal hybridization oligo parameters栏目,将引物内杂交的参数调整宽松。

图表 18高级参数调整
引物设计结果:

图表 19第一段

图表 20第二段

图表 21第三段

图表 22第四段
第一段:
| · | Sequence (5’->3’) | Template strand | Length | Start | Stop | Tm | GC% | Self complementarity | Self 3’ complementarity |
|---|---|---|---|---|---|---|---|---|---|
| Forward primer | TCGTACCTACCCTCTGTTCGT | Plus | 21 | 164 | 184 | 60 | 52.38 | 4 | 0 |
| Reverse primer | GGGCTGCACTCAGTAAGACT | Minus | 20 | 2918 | 2899 | 59.39 | 55 | 5 | 3 |
第二段:
| · | Sequence (5’->3’) | Template strand | Length | Start | Stop | Tm | GC% | Self complementarity | Self 3’ complementarity |
|---|---|---|---|---|---|---|---|---|---|
| Forward primer | CCAAGTGCCCCAACTTAGCA | Plus | 20 | 849 | 868 | 60.54 | 55 | 4 | 0 |
| Reverse primer | GGGCTGCACTCAGTAAGACT | Minus | 20 | 2918 | 2899 | 59.39 | 55 | 5 | 3 |
第三段:
| · | Sequence (5’->3’) | Template strand | Length | Start | Stop | Tm | GC% | Self complementarity | Self 3’ complementarity |
|---|---|---|---|---|---|---|---|---|---|
| Forward primer | CCAGGCGTCACTAACACAGG | Plus | 20 | 1522 | 1541 | 60.67 | 60 | 3 | 1 |
| Reverse primer | GTTCCGGGAGCGAAGTCAG | Minus | 19 | 2874 | 2856 | 60.45 | 63.16 | 5 | 1 |
第四段:
| · | Sequence (5’->3’) | Template strand | Length | Start | Stop | Tm | GC% | Self complementarity | Self 3’ complementarity |
|---|---|---|---|---|---|---|---|---|---|
| Forward primer | CGAGGGCCCCTAACAGAAAA | Plus | 20 | 2405 | 2424 | 59.67 | 55 | 7 | 0 |
| Reverse primer | CTGGGGCTGCACTCAGTAAG | Minus | 20 | 2921 | 2902 | 60.39 | 60 | 5 | 3 |
NEBcutter酶切位点分析
使用NEBcutter分析该启动子序列,为了更加全面,查找全部的特异性位点Enzymes to use: All specificities
http://nc2.neb.com/NEBcutter2/
保存没有酶切位点“0 cutters”的核酸内切酶数据,见附录。

图表 23NEBcutter结果
pGL4.17载体
查询pGL4.17的载体数据,获得酶切信息。查询关键词:promega pGL4.17。

图表 24pGL4.17载体信息
其中SfiI、Acc65I、KpnI、SacI、NheI、XhoI、EcoRV、BglII、HindIII这九个酶都属于在promoter内部没有酶切位点的,这些都可以选用。
引物设计
从上面九个酶中随便选两个(真实情况要考虑到切割率等问题)
选择KpnI和SacI,下面是酶切位点和保护碱基对应表,KpnI选两个保护碱基。

可以看出来酶切位点序列在反向互补以后和原序列相同,直接把这段序列加在引物前面就成。
最后按照“保护碱基+酶切序列+PCR引物”的顺序,设计用于可以转到pGL4.17载体的引物。
| · | 164…184–2918…2899 | 849…868–2918…2899 | 1522…1541–2874…2856 | 2405…2424–2921…2902 |
|---|---|---|---|---|
| Forward Primer | CGAGCTCTCGTACCTACCCTCTGTTCGT | CGAGCTCCCAAGTGCCCCAACTTAGCA | CGAGCTCCCAGGCGTCACTAACACAGG | CGAGCTCCGAGGGCCCCTAACAGAAAA |
| Reverse Primer | GGGGTACCGGGCTGCACTCAGTAAGACT | GGGGTACCGGGCTGCACTCAGTAAGACT | GGGGTACCGTTCCGGGAGCGAAGTCAG | GGGGTACCCTGGGGCTGCACTCAGTAAG |
后续实验流程
接下来,用这四组引物,把四个promoter区域PCR出来,顺带PCR出来的还有保护碱基和酶切序列,导入pGL4.17,用双荧光素酶报告系统看看哪儿个promoter活性最高,大概会出来下面这种图,后面那张图明显说明promoter3活性最高。


再接下来,还可以用TFSEARCH,TFBSs,TRED这样的转录因子预测软件(上面也做了几个预测),看看活性最高的那段区域和哪儿些转录因子相关,或者用pubmed查查看文献,ENCODE,TRANSFAC等数据库,查找这个基因启动子区域的转录因子信息。
接下来是编程练习部分
HMM模型
TransFac是转录因子数据库,但是好像需要注册才能下载模型的矩阵。
从EPD真核生物启动子数据库下载脊椎动物TATA-box的矩阵(共计12位碱基)。
网址http://epd.vital-it.ch/promoter_elements.php
利用该矩阵建立打分模型,对上面谷胱甘肽硫转移酶M1(GSTM1)的启动子序列进行分析,具体代码见附录。
打分值:每次取出12bp序列计算,依次计算每位碱基所占比例,再累乘得到分值(由于数值太小,分值皆除以最大分值)

图表 25打分值统计图
P值计算:使用bootstrap方法,将12bp序列打乱1000次,再按照上述方法计算分值,如果1000次内有n次分值高于“打乱之前的分值”,则p值为n/1000

图表 26 p值统计图
看的出来,整段promoter区域大部分分值都为0,p值为1。之前在线预测软件中的200,400,1200,2570这四个位置,此处也能预测出来,效果还可以。
附录
”0 cutters”核酸内切酶
| Col1 | Col2 | Col3 |
|---|---|---|
| 1 | AatII | GACGTC |
| 2 | AbaCIII | CTATCAV |
| 3 | AbsI | CCTCGAGG |
| 4 | Acc65I | GGTACC |
| 5 | Acc65V | GACGCA |
| 6 | AclI | AACGTT |
| 7 | AfeI | AGCGCT |
| 8 | AflII | CTTAAG |
| 9 | AflIII | ACRYGT |
| 10 | AhyRBAHI | GCYYGAC |
| 11 | AjuI | (N)5(N)7GAA(N)7TTGG(N)6(N)5 |
| 12 | AleI | CACNNNNGTG |
| 13 | AloI | (N)5(N)7GAAC(N)6TCC(N)7(N)5 |
| 14 | AlwFI | GAAAY(N)5RTG |
| 15 | ApyPI | ATCGAC(N)18NN |
| 16 | AscI | GGCGCGCC |
| 17 | AseI | ATTAAT |
| 18 | AsiSI | GCGATCGC |
| 19 | AspDUT2V | GNGCAAC |
| 20 | Asu14238IV | CGTRAC |
| 21 | BaeI | (N)5(N)10ACNNNNGTAYC(N)7(N)5 |
| 22 | BamHI | GGATCC |
| 23 | BarI | (N)5(N)7GAAG(N)6TAC(N)7(N)5 |
| 24 | Bce3081I | TAGGAG |
| 25 | BceAI | ACGGC(N)12NN |
| 26 | BcgI | NN(N)10CGA(N)6TGC(N)10NN |
| 27 | BclI | TGATCA |
| 28 | BdaI | NN(N)10TGA(N)6TCA(N)10NN |
| 29 | BglII | AGATCT |
| 30 | BlpI | GCTNAGC |
| 31 | BmgBI | CACGTC |
| 32 | BmtI | GCTAGC |
| 33 | BpuJI | CCCGT |
| 34 | BsaAI | YACGTR |
| 35 | BsbI | CAACAC(N)19NN |
| 36 | BsiEI | CGRYCG |
| 37 | BsiWI | CGTACG |
| 38 | Bsp24I | (N)5(N)8GAC(N)6TGG(N)7(N)5 |
| 39 | Bsp3004IV | CCGCAT |
| 40 | Bsp460III | CGCGCAG |
| 41 | BspDI | ATCGAT |
| 42 | BsrBI | CCGCTC |
| 43 | BsrGI | TGTACA |
| 44 | BssHII | GCGCGC |
| 45 | BstAPI | GCANNNNNTGC |
| 46 | BstBI | TTCGAA |
| 47 | BstEII | GGTNACC |
| 48 | BstZ17I | GTATAC |
| 49 | Bsu3610I | GACGAG |
| 50 | BtgZI | GCGATG(N)10NNNN |
| 51 | Cal14237I | GGTTAG |
| 52 | CcrNAIII | CGACCAG |
| 53 | Cdi11397I | GCGCAG |
| 54 | Cdi81III | GCMGAAG |
| 55 | CdiI | CATCG |
| 56 | Cgl13032I | GGCGCA |
| 57 | Cgl13032II | ACGABGG |
| 58 | ClaI | ATCGAT |
| 59 | Cma23826I | CGGAAG |
| 60 | CstMI | AAGGAG(N)18NN |
| 61 | DrdI | GACNNNNNNGTC |
| 62 | EagI | CGGCCG |
| 63 | EciI | GGCGGA(N)9NN |
| 64 | Eco53kI | GAGCTC |
| 65 | EcoRV | GATATC |
| 66 | Exi27195I | GCCGAC |
| 67 | FseI | GGCCGGCC |
| 68 | FspAI | RTGCGCAY |
| 69 | FspI | TGCGCA |
| 70 | GauT27I | CGCGCAGG |
| 71 | GdiII | CGGCCR |
| 72 | HindIII | AAGCTT |
| 73 | HpaI | GTTAAC |
| 74 | Hpy99I | CGWCG |
| 75 | HpyAXIV | GCGTA |
| 76 | Jma19592I | GTATNAC |
| 77 | Jma19592II | GRGCRAC |
| 78 | Kor51II | RTCGAG |
| 79 | KpnI | GGTACC |
| 80 | Lmo370I | AGCGCCG |
| 81 | Lsp6406VI | CRAGCAC |
| 82 | Maf25II | CACGCAG |
| 83 | MaqI | CRTTGAC(N)19NN |
| 84 | MauBI | CGCGCGCG |
| 85 | MkaDII | GAGAYGT |
| 86 | MluI | ACGCGT |
| 87 | MreI | CGCCGGCG |
| 88 | MslI | CAYNNNNRTG |
| 89 | MteI | GCGCNGCGC |
| 90 | NaeI | GCCGGC |
| 91 | Nbr128II | ACCGAC |
| 92 | NgoMIV | GCCGGC |
| 93 | NhaXI | CAAGRAG |
| 94 | NheI | GCTAGC |
| 95 | NotI | GCGGCCGC |
| 96 | NpeUS61II | GATCGAC |
| 97 | NruI | TCGCGA |
| 98 | PacI | TTAATTAA |
| 99 | PaeR7I | CTCGAG |
| 100 | Pal408I | CCRTGAG |
| 101 | PciI | ACATGT |
| 102 | PcsI | WCGNNNNNNNCGW |
| 103 | Pfl1108I | TCGTAG |
| 104 | PflFI | GACNNNGTC |
| 105 | PflMI | CCANNNNNTGG |
| 106 | PlaDI | CATCAG(N)19NN |
| 107 | PliMI | CGCCGAC |
| 108 | PmeI | GTTTAAAC |
| 109 | PmlI | CACGTG |
| 110 | PpiI | (N)5(N)7GAAC(N)5CTC(N)8(N)5 |
| 111 | PshAI | GACNNNNGTC |
| 112 | PsiI | TTATAA |
| 113 | PspXI | VCTCGAGB |
| 114 | PsrI | (N)5(N)7GAAC(N)6TAC(N)7(N)5 |
| 115 | Pst273I | GATCGAG |
| 116 | PvuI | CGATCG |
| 117 | RceI | CATCGAC(N)18NN |
| 118 | RdeGBI | CCGCAG |
| 119 | RpaB5I | CGRGGAC(N)18NN |
| 120 | RpaBI | CCCGCAG(N)18NN |
| 121 | RpaI | GTYGGAG(N)9NN |
| 122 | RpaTI | GRTGGAG |
| 123 | Rsp008IV | ACGCAG |
| 124 | RspPBTS2III | CTTCGAG |
| 125 | RsrII | CGGWCCG |
| 126 | SacI | GAGCTC |
| 127 | Saf8902III | CAATNAG |
| 128 | SalI | GTCGAC |
| 129 | SbfI | CCTGCAGG |
| 130 | SexAI | ACCWGGT |
| 131 | SfiI | GGCCNNNNNGGCC |
| 132 | SgrAI | CRCCGGYG |
| 133 | SgrDI | CGTCGACG |
| 134 | SnaBI | TACGTA |
| 135 | SpeI | ACTAGT |
| 136 | SphI | GCATGC |
| 137 | SpnRII | TCGAG |
| 138 | SrfI | GCCCGGGC |
| 139 | SsmI | CTGATG |
| 140 | Ssp714II | CGCAGCG |
| 141 | SstE37I | CGAAGAC(N)18NN |
| 142 | Sth20745III | GGACGAC |
| 143 | SwaI | ATTTAAAT |
| 144 | TaqIII | CACCCA(N)9NN |
| 145 | TspARh3I | GRACGAC |
| 146 | TssI | GAGNNNCTC |
| 147 | Tth111I | GACNNNGTC |
| 148 | UbaF12I | CTACNNNGTC |
| 149 | UbaF13I | GAG(N)6CTGG |
| 150 | UbaF14I | CCA(N)5TCG |
| 151 | UbaF9I | TAC(N)5RTGT |
| 152 | UbaPI | CGAACG |
| 153 | Xca85IV | TACGAG |
| 154 | XhoI | CTCGAG |
| 155 | ZraI | GACGTC |
模型矩阵
从EPD下载,保存为data.txt
http://epd.vital-it.ch/promoter_elements.php
| Position | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| A | 17.7 | 19.3 | 6.6 | 83.4 | 0 | 95 | 72.3 | 94.2 | 53.3 | 29.3 | 17.7 | 22.7 |
| C | 21.1 | 36.1 | 14.8 | 0 | 0 | 0 | 0 | 0 | 0 | 9 | 32.5 | 33 |
| G | 29 | 36.4 | 6.8 | 0 | 0 | 0 | 0 | 5.8 | 20.1 | 51.2 | 37.7 | 33.2 |
| T | 32.2 | 8.2 | 71.8 | 16.6 | 100 | 5 | 27.7 | 0 | 26.6 | 10.5 | 12.1 | 11.1 |
R代码
setwd("G:/AllShare/genomicsHomework/HMMmodel")
hmmmodel<- read.table("data.txt",header = TRUE)
rownames(hmmmodel) <- hmmmodel$Position
hmmmodel <- hmmmodel[,-1]
hmmmodel <- hmmmodel/100
hmmmodel <- t(hmmmodel)
library(seqinr)
promoter<- read.fasta(file = "promoter.fa")
seqmatrix<- as.matrix(promoter$`NC_000001.11:109684796-109687800`)
# 转成大写字母
seqmatrix<- toupper(seqmatrix)
maxseq<- strsplit("TGTATAAAAGGG",split = "")[[1]]
# 计算打分值
computeScore <- function(seq){
score <- 1
for(i in 1:length(seq)){
score <- score*hmmmodel[i,seq[i]]
}
return(score)
}
# 使用bootstrap方法,计算p值
bootstrap<- function(seq){
flag <- 0
for(i in 1:1000){
tmp <- sample(seq)
score<- computeScore(tmp)
if(score>=computeScore(seq)){
flag <- flag+1
}
}
return(flag/1000)
}
maxscore<- computeScore(maxseq)
bootstrap(maxseq)
scorevector <- c()
pvaluevector <- c()
for(i in 1:(length(seqmatrix)-11)){
tmp<- seqmatrix[i:(i+11)]
score <- computeScore(tmp)
pvalue <- bootstrap(tmp)
scorevector <- c(scorevector,score)
pvaluevector <- c(pvaluevector,pvalue)
}
result <- data.frame(scorevector,pvaluevector)
result$position <- 1:(length(promoter$`NC_000001.11:109684796-109687800`)-11)
colnames(result) <- c("score","pvalue","position")
result$score <- result$score/maxscore
library(ggplot2)
ggplot(result, aes(x=position, y=score)) +
geom_line() +
geom_point(size=4, shape=20) +
labs(title="score by HMM model")+
theme(plot.title = element_text(hjust = 0.5))
ggsave("HMM模型打分图.pdf")
ggplot(result, aes(x=position, y=pvalue)) +
geom_line() +
geom_point(size=4, shape=20) +
geom_hline(aes(yintercept = 0.05),colour="red",linetype="dashed")+
labs(title="p value by HMM model")+
scale_x_continuous(breaks = c(0,200,400,1030,1200,1632,2570,2700))+
theme(plot.title = element_text(hjust = 0.5))
ggsave("HMM模型p值图.pdf")
序列
Promoter序列
>NC_000001.11:109684796-109687800 Homo sapiens chromosome 1, GRCh38.p7 Primary Assembly
TCTGCTCTTGAACCCTGTGTTCTGTTGTTTAAGATGTTTATCAAGACAATATGTGCACCACTGAACATAG
ACCCTTATCAGGAGTTCTACTTTTGCCTTTGTCCTGTTTCCTCAGAAGCATGTGATCTTTGTTCTGCTTT
TTGCCCTTTAAAGCATGTGATCTTCGTACCTACCCTCTGTTCGTACACCACCACCCCTTTTGCAATCCTT
AATAAAAACTTGCTGGTTTTGAGGCTCGGGCAGGCATCATGGTCCTACCGATATGTGATGTCACCCCTGG
CGGCCCAGCTGTAAAATTCTTCTCTTTGTACTCTTTCTCTTTATTTCTCAGCTGGCTGACACTTATGGAA
AATAGAAAGAACCTACGTTGAAATATTGGGGGCAGGTTCCCCAATAGCCTTGCTGAGGAAATTAAATTTA
TGTTCAAGTGCTATTTCTTTATGGCACCAAGGAACAAGTATTTCAAACAATACTAATGTAACAGTACTGG
TTCTATGTGTTTCAAAATTATTATTCTCATGAGTGTTAGCTTTCTTAAAAAATCGTTTTTTTTTTCAATT
GGATCTAGACATCTTATCTTTCACAGCTCAAGACGGATTAACTCAGAATCATAAACTCTTAATGCATAAT
GAGAAATATAATGTTTCCTAGGGCCAGGCACTTGTGTCTGTGCTGGTGCTATTGCCTCAATGCAGGAAAA
TCTATGTAAGAGTTCACTGTGAGGCCAAAACTGCTTCCTAAACATGGATACCTGCCAGGTATCTGAGCTG
GGAGTACTGCCCAGGTCTGGATGGGCGGGGAGTGTTTGCAACAAGGACTGTGCCTTGCCAGCCTCAGTGA
CACAGTGTCCAAGTGCCCCAACTTAGCAGCCACCTGCTGACCACCTGATTTCTGTGGCCTAATAGGGATG
TGATGAAGTCTACCTGTTTACTCAACCCCAAACCACACATTATCCAGGTGGTTTGAAACTTTTTTGATAT
ACTGGGTTCATCCTCTGGAGTCCTAACAATGTTTTAGCTAATTTACAAAAAACAAAACAAAACAAAACAA
AACAAAACAAAACAAAAAAACTACTTTTTTTGCAGCACAACAGCCTGGTTTACATTGCAAAATGATTTCT
CATTAAAGGTCTATCATCTATTTCCATATGTCCATTATTATTTGCAATATCCTTTAAAGCAGTCAACCCC
AGGCTAATCCATTGCACAACTCTTTTGAAAGTCTTCCTTCTACCTTGAAAGAAGAAAGTTGGCAGGTTGG
ACATTGTTCTCGTGGAGGTTGTACCATGGGTCACATATCACGGTGTGACTTCAAAGGCCACTGGAGCCAC
CGTCTCATACTGAAGAACACACATGGGTCAGGAGCCAGGTCCAGGTCCGGAATGGTGGATCTGGAGAGGG
GAGGGTCCCTGCCTGTGGTCCTGTGGGGAGCCCTCAGGCTCCTCTCTGGCCACCATCCTCTGACCTCCCT
CCTCAGCAGGACAGGGTTCTGGCTTCTCTGAGGGACAGGTTCTGTGGCAGGCCAGGCGTCACTAACACAG
GCCTCCATAACAACTGTTTCAGTACTGACTGAGTGGTGAAGTTAAATATTAAAAGCTGAAAAAAGCCAGT
ACCTTTATACAGAGGCTGGATGTAACAAAAGCCCACCAAGAGTTTTGCTTAGGCCTTTCCTGGGCCTTAA
AGCATGACAAAACAATGAAGGAATTCTTAACAGGACCTATTTAGAATTAAACAAGTTTTATTGTGAGTCT
GAAGAAACTCCCCAGGCCTCCACAAACAAGTTTATTGGGCGTCTGAAGGAACTCCCCAAACCTCCGTGAT
TTAGCAGGAGACAAGATAAGGGTAATCATCCCCCGCACCTGGACCCATTTAGATTAAATAAATAGACTGA
GGCTCCAGAATAAGGTCCTCAGGACCCAGACCTCAGTTACAGATTAAAGAAGTTAATCACTTATGTCTTT
AGATGAATGCACACTTACTTGTAGACATATACCTTAGAAGGTATATATGCTCTGGAAAACTTTGTAATAT
TGAGTTGGTCTGGTGGTAATTTCTAGGCCTTCTCCCTGTTACCGGTTGCAGAAATAAAACCTCTCTTCCT
CCCCATTTGATCTGCATCTCGTTATTGGGCCTAGAGAAATAGCAGCCGGACCCTCAGTTTGGTCCGGGAA
GTTCTTCCATCCTCCCTCGCCTGCTCTCTGTGGCCACTGCACTCACTGTTGCTGTTGCTGTTCCGGTCTC
TGTGAGGTTCACCTAGTGGACTGGCTGGACATTTCTAGGGGGCACCTCAGATACCTCACCAACTTGCTGG
ATCTGATCCTTGGATTTCGATTCATAAATTGTGCCAAAATACGAAGTGGCTAATTTACACAGTACTTAGC
CAGATGACCGAAGGACTCAGTACCCGAGGGCCCCTAACAGAAAACACAGACCACATTTCCTTTACTCTGG
CCCTTTTCCTGGGGGTCCTTCCTATACCACTGACACTGTTCCTGTGTAGGCGGGGCTAGAGGGGAGACTA
AGCCCTGGGAGTAGCTTTCGGATCAGAGGAAGTCCTGCTCTTACAGTGACAGGGGCTGAATTAAATTCCC
AGGTTGGGGCCACCACTTTTTAGTCTGACCCCTGCAGCCGGAGTCTCCCAGAGCCCTTGGGAACTCGGCA
GCGGAGAGAAGGCTGAGGGACACCGCGGGCAGGGAGGAGAAGGGAGAAGAGCTTTGCTCCGTTAGGATCT
GGCTGGTGTCTCAAGCGCACAGCCAAGTCGCTGTGGACCTAGCAAGGGCTGGATGGACTCGTGGAGCCTC
AGGGCTGGGTAGGGAAGCTGGCGAGGCCGAGCCCCGCCTTGGGCTTCTGGGCGCCCTGACTTCGCTCCCG
GAACCCTCGGGCCTGGGAGGCGGGAGGAAGTCTTACTGAGTGCAGCCCCAGGCGCCCTCTCCCGGGCCTC
CAGAATGGCGCCTTTCGGGTTGTGGCGGGCCGAGGGGCGGGGTCGCAGCAAGGCCCCGCCTGTCC
转载自原文链接, 如需删除请联系管理员。
原文链接:启动子的分析和预测,转载请注明来源!
